Bioinformatique
Rapports de bioinformatique

Parcourez nos rapports de bioinformatique ci-dessous.
Découvrez notre rapport (assemblage de-novo)
Découvrez notre rapport (expression différentielle)
Rapport bioinformatique
-
Figures et tableaux interactifs
-
Informations enrichies sur les données
-
Exploration des données à partir du rapport
-
Zoom et filtrage des données
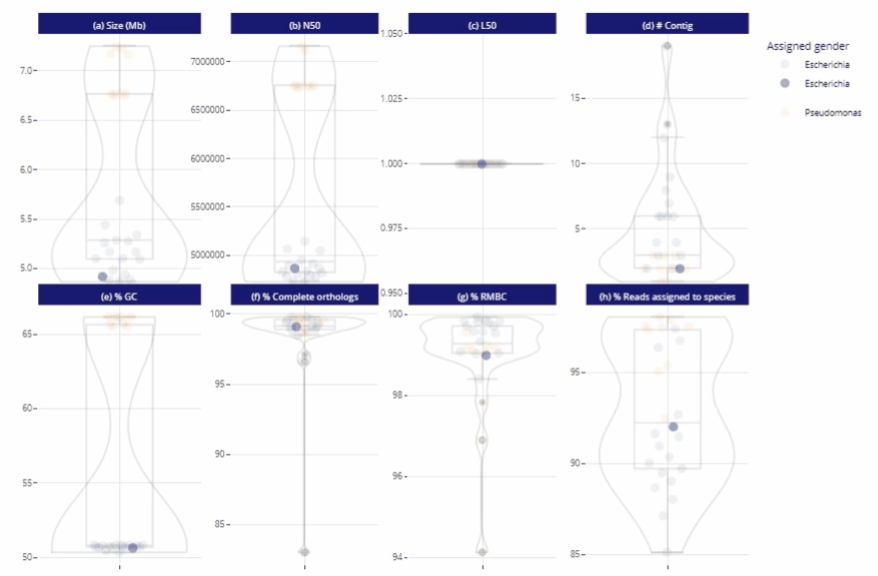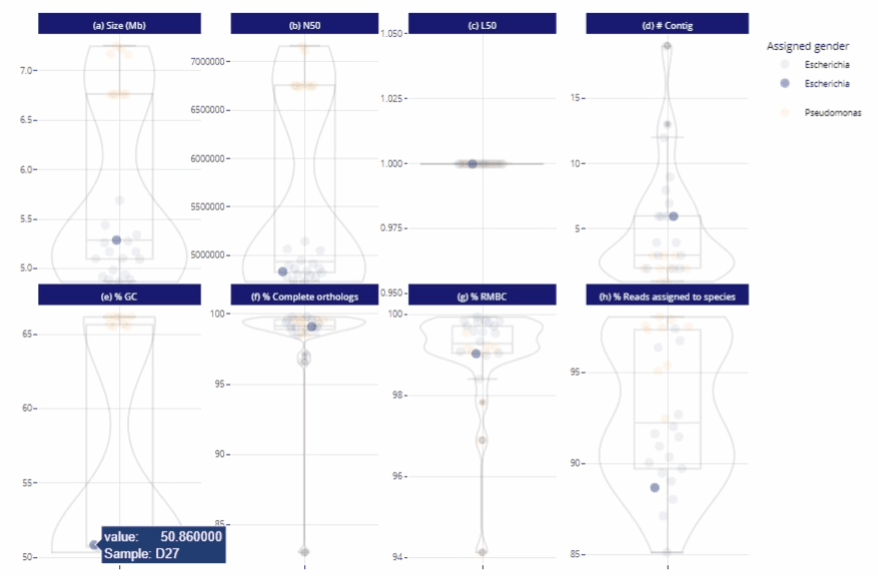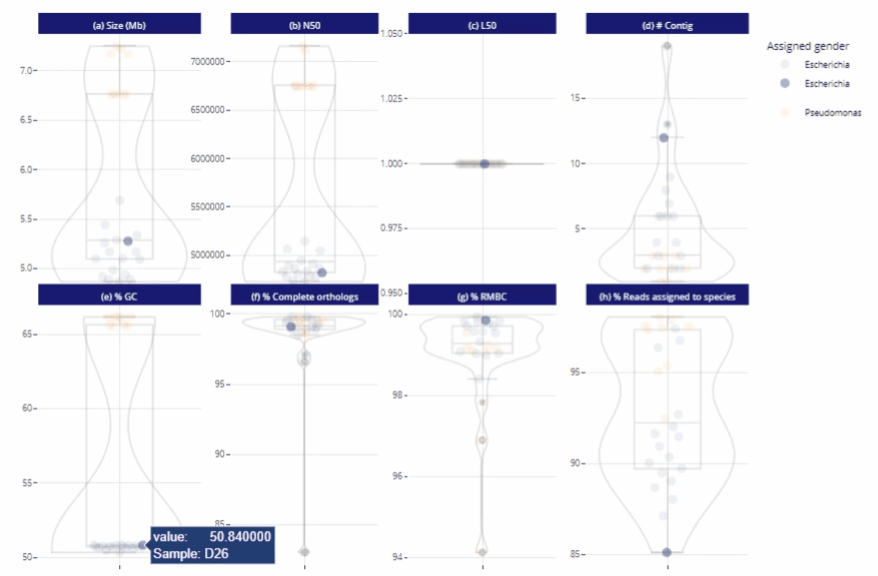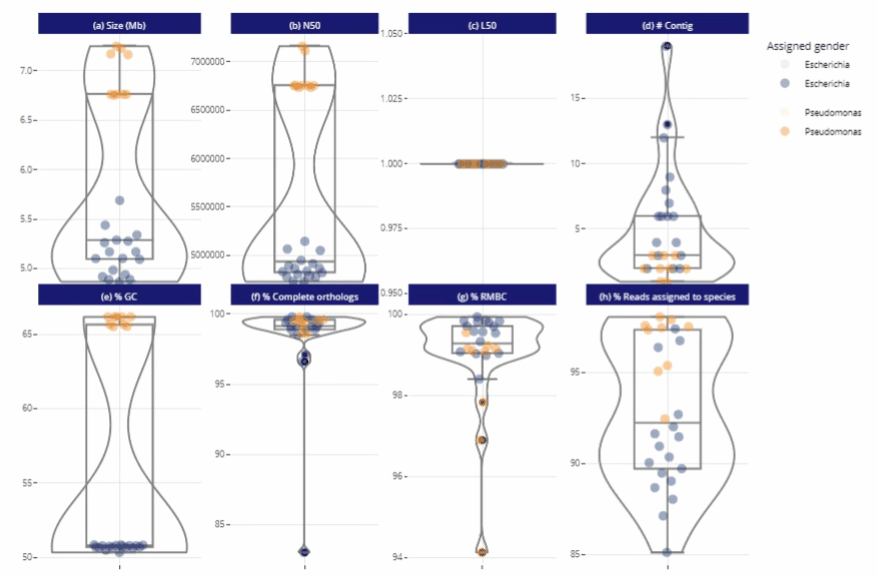
Différents niveaux de lecture
DU NÉOPHYTE A L'EXPERT EN BIOINFORMATIQUE
-
Première page contenant un résumé graphique et écrit
-
Détail de chaque étape de l'analyse
-
Résultats clés mis en avant

Adapté a toute taille de projet
-
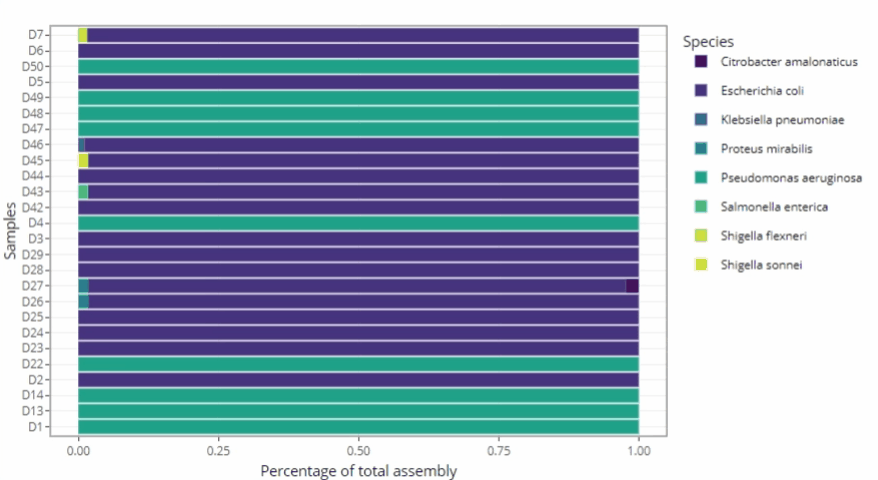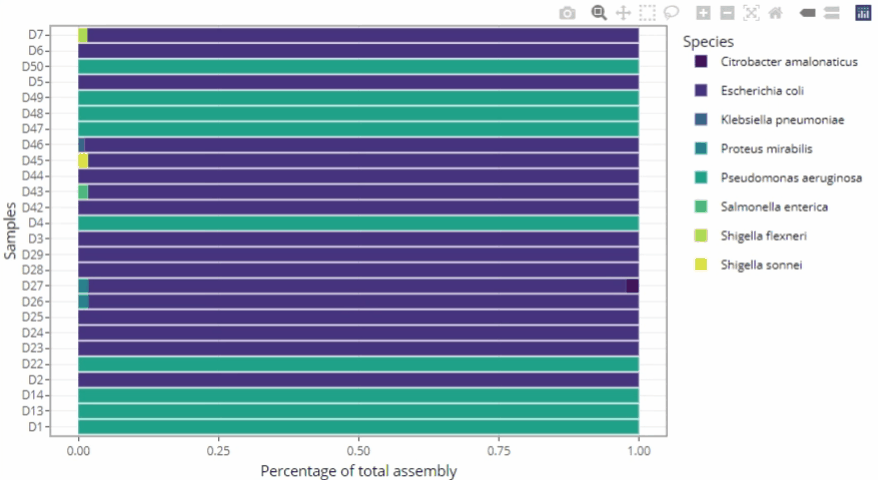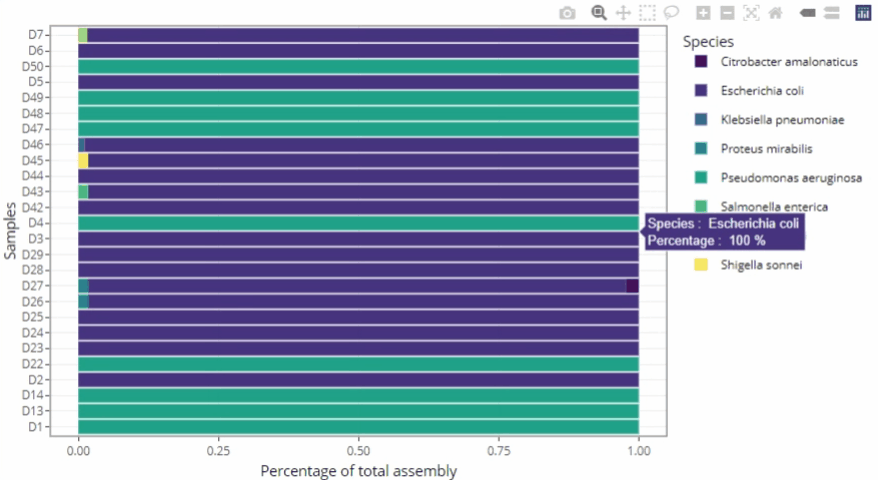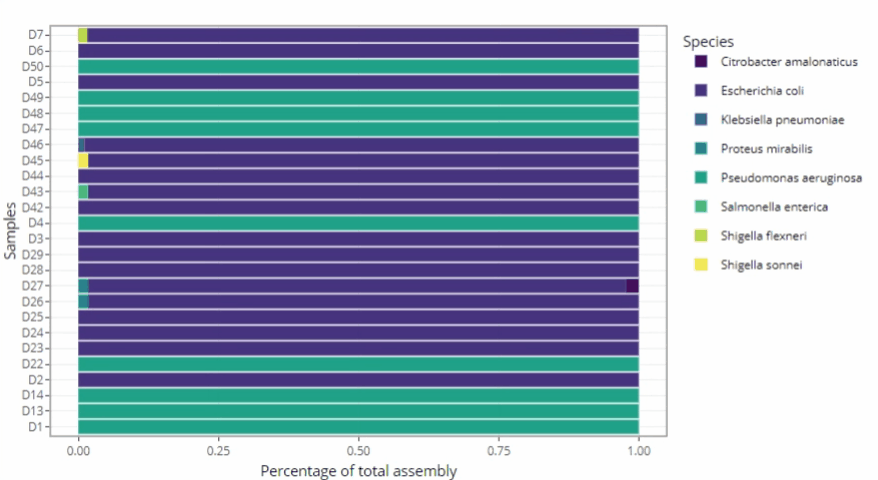
Représentations graphiques s'adaptant automatiquement au nombre d'échantillons
-
Clarté et lisibilité pour un seul échantillon comme pour des centaines.
Méthodologie « ready-to-publish »
-
Section détaillée sur le matériel et les méthodes
-
Transparence sur les outils et méthodes utilisés
-
Références et sources déjà préformatées pour la publication
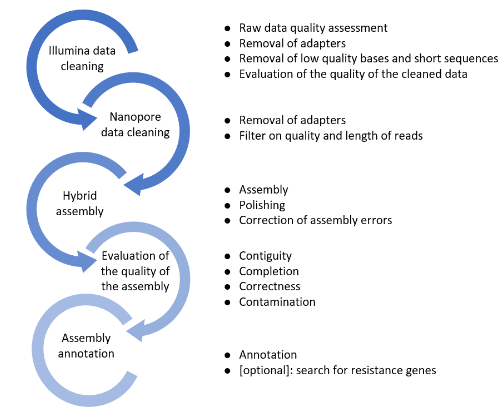
Comparaison de génomes et détection de mutations
L’un des grands défis de la génomique est de pouvoir lier le phénotype avec l’information génétique disponible. La détection de mutations et la comparaison de génomes complets permet de dépasser le stade de l’observation génomique, pour donner un sens aux données de séquençage, pour faire progresser vos recherches.
Applications de la comparaison de génomes et de la détection des mutations
L’analyse comparée des génomes et la détection de mutations sont mobilisées pour repérer les gènes et les mutations d’intérêt, par rapport à un phénotype moléculaire ou cellulaire : antibiorésistance, action enzymatique, etc.
Ces deux approches permettent, par exemple, de mettre en place de nouveaux critères de sélection génomique pour l’agriculture, de repérer des cibles thérapeutiques, de sélectionner des souches spécifiques, de découvrir de nouveaux biomarqueurs, etc.
L’offre GenoScreen - Détection de mutation
La détection de mutations (SNP ou INDEL) est réalisée par comparaison de séquences proches (ex : génomes/transcriptomes de deux organismes appartenant à la même espèce) afin de repérer des mutations d'intérêt.
Cette comparaison passe par un mapping des séquences obtenues sur une séquence de référence. La portion de génome de référence couverte doit alors être similaire pour les deux séquences comparées.
L’avantage GenoScreen
- Traitement spécifique des informations de séquençage, pour améliorer qualité et lisibilité.
- Une annotation détaillée des gènes et séquences d’intérêt
- Algorithmes précis d’analyses des alignements sur les séquences de référence
- Capacité à comparer des génomes complets
Bioinformatique - Donner une réponse claire à vos hypothèses de recherche
GenoScreen développe une large gamme de services bioinformatiques, notamment destinée à l’interprétation des données issues du Next Generation Sequencing (NGS). Les algorithmes GenoScreen offrent des réponses claires et précises à tout type de problématique génomique.
Des outils exclusifs d’interprétation et d’analyses
Nos services de bioinformatique sont nourris en permanence par les résultats de nos programmes de R&D. GenoScreen dispose d’outils de post-traitement des données brutes du séquençage qui garantissent la plus haute qualité d’information. De nombreux algorithmes spécifiques sont développés en collaboration avec nos experts de la génomique, pour accélérer l’interprétation des résultats et apporter une information scientifique plus lisible et plus riche.
Expression différentielle des gènes - Comprendre l’influence génétique sur le comportement cellulaire
L’expression génique est un champ d’exploration majeur pour mieux comprendre les mécanismes d’action impliqués dans le comportement cellulaire. GenoScreen propose des services complets d’analyse génomique différentielle pour vous aider à progresser dans le repérage de biomarqueurs d’intérêt.
Les applications de l'analyse génomique différentielle
Les technologies RNA-Seq permettent l’analyse de l'expression différentielle de l’ensemble du génome d’un organisme et la comparaison du niveau d'expression de gènes entre deux conditions, afin de rechercher des biomarqueurs d’intérêt :
- Comparaison entre tissus sains et pathologiques
- Comparaison de comportement cellulaire entre deux environnements différents (nutrition, stress, pollution, etc.)
- Comparaison du comportement cellulaire à différents stades de développement biologique
Cette approche exhaustive comporte plusieurs avantages :
- Pas de connaissance préalable requise sur l'organisme étudié
- Analyse de l'ensemble du transcriptome sans a priori sur les gènes d'intérêt
- Possibilité d'utiliser les données de séquençage pour générer un transcriptome de novo
- Analyse des NATs
L’offre GenoScreen - Analyse génomique différencielle
Nos équipes proposent un accompagnement complet et personnalisé :
- Assistance au design de l’étude
- Purification de l’ARN
- Préparation des librairies et du séquençage
- Pool et séquençage
- Analyse bioinformatique
L’avantage GenoScreen
- Prestation complète, du design à l’analyse bioinformatique des résultats
- Protocoles optimisés pour maximiser sensibilité, fiabilité et reproductibilité de l’analyse
- Rapport d’analyse précis et synthétique, détaillant les différences les plus significatives
Détection de mutations
Objectifs
La détection de mutations (SNP/INDEL) permet de comparer des séquences proches (ex : génomes/transcriptomes de 2 organismes appartenant à la même espèce) afin de repérer des mutations d'intérêt.
Mapping
Le mapping est un alignement de séquences courtes (typiquement des reads issus de séquençage Illumina) sur une référence au format FASTA. L’étape de mapping permet de réaligner les reads sur la référence pour détecter et localiser les mutations sur le génome.
Exemples d’applications
- Médicale : recherche de mutations impliquées dans des maladies génétiques, tests de dépistage de certains cancers
- Pharmaceutique : prédiction de réponse à un traitement
- Agronomique : sélection de lignées
- construction de cartes génétiques, études de phylogénie
Outils utilisés
- Bowtie2 : Réalignement des reads sur la référence
- Suite logicielle samtools : Filtrage des alignements et détections des mutations
Pré-requis
- Pour chaque échantillon : données de séquençage au format FASTQ avec une profondeur minimum allant de 30X pour la détection de SNP homozygotes à 50X si on souhaite également détecter les SNP hétérozygotes.
Il est généralement conseillé de travailler avec une profondeur de 50X : http://bmcbioinformatics.biomedcentral.com/articles/10.1186/1471-2105-15-247 - Séquences de références au format FASTA (transcriptome, génome...). Le génome ou transcriptome de référence peut être généré par un assemblage de novo des reads.
Rendu client
Fichiers de comparaison au format VCF ou CSV.
Délais de réalisation
Paramètres à prendre en compte
- Taille et nombre de séquences de la référence
- Nombre d'échantillons.
Assemblage - Reconstituer fidèlement les données génomiques
Les performances des technologies NGS reposent en grande partie sur les méthodologies et les capacités bioinformatiques de reconstituer l’intégralité des séquences visées (contigs ou scaffolds) à partir d’un très grand nombre de fragments de génomes ou transcriptomes.
La première tâche est d’ordonner et d’assembler ces fragments d’information en s’appuyant sur des génomes de référence. Le but est d’identifier les variations avec le standard, puis, dans un second temps, d’en comprendre le rôle et l’influence (commentaire, interprétation). Pour cela, nos bioinformaticiens font appel à des algorithmes sous forme de processus semi automatisés. Cela nécessite des capacités de calcul et d’analytique considérables, de sorte que la génomique est aujourd’hui l’un des premiers champs d’application du calcul haute performance (HPC) et du Big Data.
Outre la puissance de calcul, le stockage des données constitue un autre enjeu majeur de la génomique, non seulement en termes de volumétrie et de performances, mais aussi de sécurité. En effet, les données sont hautement sensibles et confidentielles.
Les applications de l'assemblage
L’assemblage permet de reconstituer un génome complet à partir des nouvelles techniques de séquençage, soit en s’appuyant sur des génomes de référence, soit dans une démarche de novo.
Cette reconstruction donne une image plus complète et plus détaillée des données génomiques, pour en faciliter l’interprétation.
L’offre GenoScreen - Assemblage
Pour obtenir les meilleurs résultats, GenoScreen associe les méthodes les plus adaptées à chaque problématique :
- L’assemblage de novo, qui assemble les reads issus du séquençage (génome ou transcriptome) en séquences plus longues (contigs ou scaffolds) avec l'objectif d’obtenir une séquence par chromosome ou transcrit unique.
- Le mapping, qui aligne les fragments courts sur une séquence de référence au format FASTA
Nos méthodes d’assemblage reposent sur plusieurs algorithmes utilisés par nos équipes pour optimiser la longueur des contigs assemblés et réduire au minimum les erreurs d’assemblage.
L’avantage GenoScreen
Un taux d’erreur limité grâce à une méthodologie d’assemblage de novo exclusive
Demander un devis
Annotation - Comprendre le génome
Les annotations génomiques visent à associer le séquençage d’un génome ou d’un transcriptome à une information biologique exploitable. À partir d’assemblages de haute qualité, GenoScreen fournit des annotations précises qui sont indispensables à la compréhension des fonctions biologiques des organismes.
Les applications des annotations génomiques
En reliant les données de séquençage et les connaissances déjà acquises sur un génome, les annotations approfondissent la connaissance d’un génome.
Le but est d’identifier les variations avec le standard puis, dans un second temps, d’en comprendre le rôle et l’influence. Les annotations donnent des indications pour comprendre les évolutions d’un organisme ou de son expression génique (pathologie, production agroalimentaire…).
L’offre GenoScreen - Annotations génomiques
- L'annotation structurale permet de rechercher et de localiser les régions codantes d’une séquence pour prédire la position des gènes (Open Read Frames - ORFs)
- L’annotation fonctionnelle compare par alignement les séquences obtenues aux banques de données constituées de séquences déjà annotées. Elle permet de rechercher la présence de motifs spécifiques à certaines fonctions biologiques.
L’avantage GenoScreen
- Recherche exhaustive dans les bases de référence de génomique fonctionnelle (UniProt / GeneOntology, etc.)
- Capacité importante de calcul HPC
- Technologies avancées de Big Data
- Excellente connaissance de la génomique fonctionnelle microbienne